Virus ARN monocatenario negativo
| Virus ARN monocatenario negativo | ||
|---|---|---|
 | ||
| Taxonomía | ||
| Clasificación de Baltimore | ||
| Grupo: | V (Virus ARN monocatenario negativo) | |
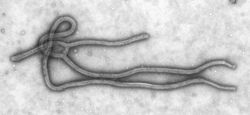
Un virus ARN monocatenario negativo (abreviado virus ARNmc- o virus (-)ssRNA en inglés) es un virus que tiene ácido ribonucleico (ARN) de cadena sencilla de sentido negativo como material genético y no se replica usando ADN intermedio. Pertenecen al Grupo V de la clasificación de Baltimore.[1] Es una clase de virus del tipo ARN monocatenario, los cuales pueden clasificarse según el sentido o polaridad de su ARN en negativos o positivos. El ARN viral negativo es complementario del ARNm y por lo tanto debe convertirse en ARN positivo por una ARN polimerasa antes de la traducción. El ARN purificado de un virus negativo no es por sí mismo infeccioso puesto que necesita ser traducido en ARN positivo. Este grupo también incluye a los virus ARN monocatenarios ambisentido (abreviado virus ARNmc+- o virus (+/-)ssRNA en inglés). Entre los virus de este grupo que infectan a los humanos destacan los virus de Marburgo, Ébola, sarampión, parotiditis, rabia y gripe.
Clasificación taxonómica[editar]
La clasificación taxonómica del ICTV y complementada con otros estudios es la siguiente:[2][3]
- Filo Negarnaviricota
- Filo Arctoviricota
Multiplicación[editar]
Los virus ARN de sentido negativo utilizan una ARN polimerasa o transcriptasa para formar ARN de sentido positivo. Esto significa que el virus debe aportar la enzima ARN polimerasa puesto que ésta es dependiente del ARN. La molécula ARN de sentido positivo entonces actúa como un ARNm viral, que se traduce en proteínas por los ribosomas del huésped. Las proteínas resultantes se dedican directamente a la producción de los elementos de los nuevos viriones, tales como las proteínas de la cápsida y la ARN replicasa, que se encarga de la producción de nuevas moléculas de ARN de sentido negativo.
| Síntesis de proteínas: | ARNm- → ARNm → proteínas |
| Replicación del genoma: | ARNmc- → ARNmc+ → ARNmc- |
| Enzimas: | ARN polimerasas aportadas o codificadas por el virus: ARNmc- → ARNm, ARNmc- → ARNmc+, ARNmc+ → ARNmc- |
La multiplicación del virus comprende las siguientes etapas:[4]

- Transcripción temprana del ARN de sentido negativo por la ARN polimerasa dependiente del ARN dentro del virión y producción principalmente de ARNm subgenómico y también de ARN monocatenario positivo. Este paso se produce en el citoplasma y da lugar a la formación del complejo replicativo.
- Traducción del ARNm y producción y acumulación de las proteínas tempranas (reguladoras).
- Las proteínas reguladoras interactúan con el complejo replicativo, incentivando la producción del ARN monocatenario positivo y por lo tanto del ARN monocatenario negativo genómico.
- Transcripción tardía del ARN monocatenario negativo.
- Traducción tardía del ARN monocatenario negativo y producción y acumulación de las proteínas tardías (estructurales).
- Ensamblado de la nucleocápside y maduración. Gemación de la nucleocápside a través de la membrana celular de donde se obtiene la envoltura viral.
El genoma puede ser no segmentado con varios ORF (Filoviridae, Paramyxoviridae y Rhabdoviridae), dos segmentos ambisentido (Arenaviridae), tres segmentos, ocasionalmente ambisentido (Bunyavirales y Tenuivirus) o de seis a ocho segmentos (Orthomyxoviridae). El tamaño del genoma está comprendido entre 10 y 30 kb. Podemos distinguir, por tanto, dos subgrupos de virus:
- Virus que contienen genomas no segmentados para los cuales el primer paso en la replicación es la transcripción de la cadena negativa por la ARN polimerasa dependiente el ARN viral para formar varias cadenas de ARNm monocistrónico que codifican las distintas proteínas virales. Se forma entonces una copia del genoma de sentido positivo que sirve como plantilla para la producción del genoma negativo. La replicación tiene lugar en el citoplasma.
- Virus con genomas segmentados para los cuales la replicación se produce en el núcleo y en los que la ARN polimerasa dependiente del ARN viral produce una cadena ARNm monocistrónica a partir de cada segmento del genoma. La principal diferencia entre los dos tipos de virus es el lugar en donde se realiza la replicación.
Por huéspedes, ejemplos de taxones que infectan animales son las familias Orthomyxoviridae, Paramyxoviridae, Filoviridae, Arenaviridae, Bornaviridae, Hantaviridae, entre otras, en plantas; Aspiviridae, Fimoviridae, Tospoviridae, entre otras, en hongos; Mymonaviridae, Discoviridae, Tulasviridae, en protistas; Leishbuviridae, Yueviridae. Otros taxones en cambio infectan huéspedes distintos por cruzado, animales, plantas y hongos; Phenuiviridae, animales, plantas, hongos y protistas; Rhabdoviridae, todo tipo de microorganismos; Arctoviricota.
Galería[editar]
Véase también[editar]
Referencias[editar]
- ↑ ICTV Master Species List 2018b.v1 (Website). U.S. National Center for Biotechnology Information, National Library for Medicine, National Institutes of Health. Consultado el 30-marzo-2019.
- ↑ «Virus Taxonomy: 2019 Release» (html). International Committee on Taxonomy of Viruses (ICTV) (en inglés). October 2018. Consultado el 13 octobre 2019.
- ↑ Cryptic and abundant marine viruses at the evolutionary origins of Earth’s RNA virome. Science org.
- ↑ Charles E. Samuel (2005) Virus-Host Interaction Minireview Series: Human Immunodeficiency Virus, Hepatitis C Virus, and Influenza Virus, J. Biol. Chem., Vol. 281, Issue 13, 8305-8307.
Enlaces externos[editar]
 Wikispecies tiene un artículo sobre Virus ARN monocatenario negativo.
Wikispecies tiene un artículo sobre Virus ARN monocatenario negativo. Wikimedia Commons alberga una categoría multimedia sobre Virus ARN monocatenario negativo.
Wikimedia Commons alberga una categoría multimedia sobre Virus ARN monocatenario negativo.- ViralZone
- Replication Strategy of ss(-)RNA Viruses












